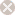2024年8月,两篇SCI文章因细胞名称错误拼写、使用问题细胞系在Pubpeer上被质疑,其中一篇文章在2024年8月8日已经被撤稿!涉及的细胞系在Cellosaurus数据库中都有相关预警信息。
2018年3月,中国三峡大学某临床医学院在DNA Cell Biol.期刊发表了一篇关于胃癌的文章。作者使用的“BSG823”和SGC-7901细胞系受到关注。Hoya camphorifolia在Pubpeer上指出,作者首先出现了细胞系名称拼写错误,“BSG823”是一株不存在的细胞系,它其实是BGC-823细胞系的错误拼写;另外,BGC-823和SGC-7901都是问题细胞。ExPASy的预警信息显示,这两株“胃癌细胞系”都已经被HELA污染(具体可参看《SGC-7901:苏州鉴达细胞株鉴定预警系列5》和《BGC-823:苏州鉴达细胞株鉴定预警系列6》)。
Pubpeer评论中所提到的Oste et al (2024)文章,此前我们公众号推文曾对其进行过解读(具体可参看《警惕!多个拼写错误的细胞名称被视为独立细胞系!》)。Oste et al (2024)这篇文章发现,大量SCI论文涉及使用NV(Non-Verifiable)细胞系名称,而且其中一部分NV细胞系名称正逐渐被视为独立的细胞系。
(图片来源:Pubpeer)
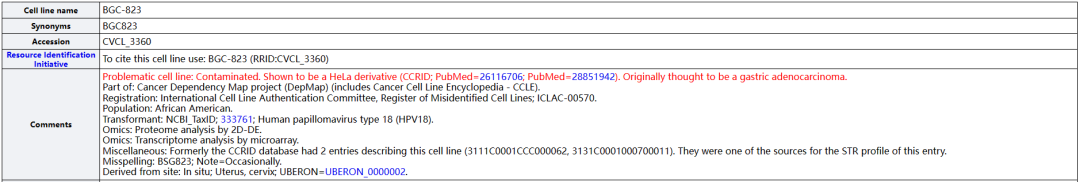
(图片来源:ExPASy)
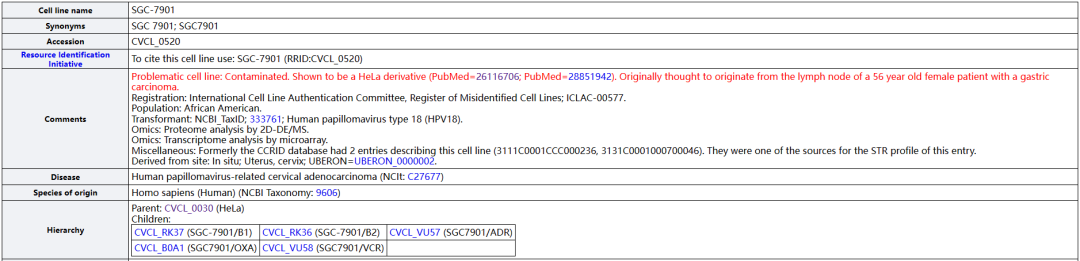
(图片来源:ExPASy)
暨南大学某附属医院2021年5月在J Gene Med.期刊发表一篇文章。作者使用的“BGC803”、“BSG823”及“GSE1”细胞系引起科研同行的注意。早在2023年3月,Amos Bairoch就在Pubpeer上指出,作者出现了细胞系名称拼写错误,“BGC803”、“BSG823”及“GSE1”都是不存在的细胞系,他们分别是MGC-803、BGC823、GES1的错误拼写!更重要的是,MGC-803和BGC823都是问题细胞系,MGC-803的STR数据是HELA与胃癌细胞的混合数据;BGC823被HELA污染。
另外,这篇文章多处实验图片也存在问题。2024年8月8日,文章被撤稿!相关撤稿信息也更新在Pubpeer网站。

(图片来源:PubPeer)

(图片来源:PubPeer)
参考文献:
1.www.jsdna.org/cell
2.http://www.jsdna.org/mobile/information/xuz
3.https://pubpeer.com/publications/06D2C83175062EB9586FA9D2B2D837
4.https://pubpeer.com/publications/5C9535F56FEE8D7FD153C7306FB80B
苏州鉴达(Genetic Testing Biotechnology, GTB)采用STR分型技术,针对人源细胞23个基因座、小鼠源细胞19个基因座(18个小鼠源基因座+1个人源基因座TH01)进行检测,3130XL型遗传分析仪进行毛细管电泳,与DSMZ、Cellosaurus、ATCC等数据库进行比对,出具权威英文报告,为全国近550家科研机构(高校、研究所、医院、公司)提供高质量服务,多家单位已利用鉴达细胞株鉴定服务在高水平期刊(如Clinical Cancer Research、Nature Commnications、Int J Cancer等)发表论文。如需了解更多鉴达生物(Genetic Testing Biotechnology, GTB)相关细胞株鉴定信息,可扫码关注官方公众号。将本文转发至朋友圈(至少保留三天)。添加微信或QQ客服后,将朋友圈截图发给微信或QQ客服,并备注单位及课题组(示例:XX大学XX课题组)。后期可有两次机会享七五折优惠,原价1200元/株。微信:18112556907、QQ:2791427499
标注优惠政策:如客户在发表的高水平SCI论文(IF>10)中标注细胞株鉴定由我公司完成,我们承诺为客户免费检测两株细胞株。格式如下:The cell lines were characterized by Genetic Testing Biotechnology Corporation (Suzhou,China) using short tandem repeat (STR) markers.
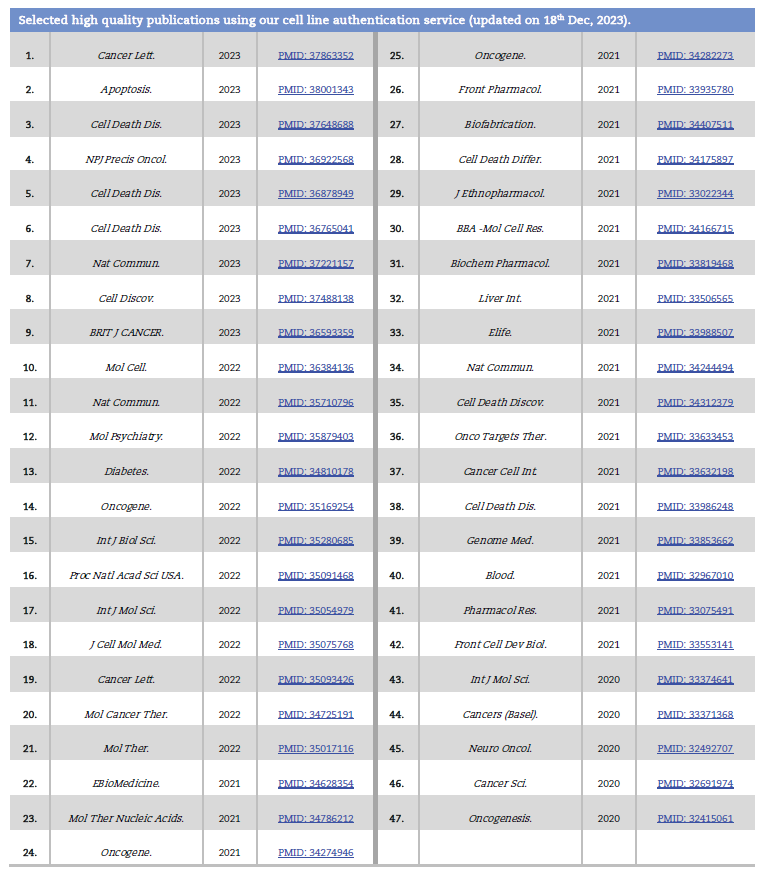
Selected high quality publications using our cell line authentication